class: center, middle, inverse, title-slide .title[ # Demeter y Euler van por una cerveza ] .subtitle[ ## Cuantificando la forma de la cebada con ATD ] .author[ ### <strong>Erik Amézquita</strong>, Michelle Quigley, Tim Ophelders <br> Elizabeth Munch, Dan Chitwood <br> Dan Koenig, Jacob Landis <br> - ] .institute[ ### Computational Mathematics, Science and Engineering <br> Michigan State University <br> - ] .date[ ### 2020-12-15 <br> - <br> Diapositivas: <code>https://bit.ly/cebada_fest</code> ] --- background-image: url("../../demat/figs/fam9_3.png") background-size: 150px background-position: 95% 5% # Cómo inició todo... - Fragmento de febrero 2016. (Énfasis propio) > Estimados Estudiantes de Licenciaturas de Demat, > > Se les hace una cordial invitación a participar como estudiantes de verano (con posibilidad de comenzar a pensar en una tesis en el futuro), en el proyecto interdisciplinario de Análisis Topológico de Datos-ATD aplicado a objetos Arqueológicos en 3D. > > Se anticipa que partes de este proyecto requerirán de elementos de **análisis funcional, análisis armónico, topología/geometría diferencial, probabilidad y estadística, homología y programación**. Algunos elementos se tendrán que aprender sobre cómo parte de la participación. -- - Fui el único que manifestó interes -- - Sabía prácticamente nada de los requisitos. --- background-image: url("../../img/endlessforms.png") background-size: 150px background-position: 89% 7% class: inverse # Ahora: Morfología botánica <div class="row"> <div class="column" style="max-width:50%"> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/oM9kAq0PBvw?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/V39K58evWlU?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> </div> <div class="column" style="max-width:50%"> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/4GBgPIEDoa0?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/qkOjHHuoUhA?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> </div> </div> <p style="font-size: 24px; text-align: right; font-family: 'Yanone Kaffeesatz'">Mira más tomografías 3D en <a href="https://www.youtube.com/@endlessforms6756">youtube.com/@endlessforms6756</a></p> --- # Análisis Topológico de Datos (ATD) <div class="row"> <div class="column" style="max-width:25%; font-size: 15px;"> <img style="padding: 25px 0 35px 0;" src="../figs/S019_L0_1.gif"> <p style="font-size: 25px; text-align: center; color: DarkRed;"> Datos </p> <ul> <li> Escanes rayos X </li> <li> Nubes de puntos </li> <li> Series de tiempo </li> <ul> </div> <div class="column" style="max-width:40%; padding: 0 25px 0 25px; font-size: 15px;"> <img src="../figs/ecc_X.gif"> <p style="font-size: 25px; text-align: center; color: DarkRed;"> Resumen Topológico </p> <ul> <li> Característica de Euler </li> <li> Diagramas de persistencia </li> <li> Mapper </li> <ul> </div> <div class="column" style="max-width:35%; font-size: 15px;"> <img src="../figs/svm_mds_ect.gif"> <p style="font-size: 25px; text-align: center; color: DarkRed;"> Análisis </p> <ul> <li> Estadística </li> <li> Aprendizaje de máquina </li> <li> Clasificación y predicción </li> <ul> </div> </div> --- background-image: url("../figs/seed.png") background-size: 400px background-position: 99% 99% # Resumen de hoy ### La charla será muy modular 1. Botánica: cebada 1. ~~Genética: Zigosidad~~ 1. Procesamiento de imágenes 1. Transformada de característica de Euler 1. Aprendizaje de máquina y resultados 1. Genética en el horizonte --- class: center, middle, inverse # 1. Botánica: Cebada --- class: inverse <div class="row"> <div class="column" style="max-width:44%"> <!-- <a href="https://www.thescottishfarmer.co.uk/arable/14364703.hybrid-barley-pays-off-for-mixed-farming-too/" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_scotland.jpg"></a> --> <a href="https://cereal.interreg-npa.eu/subsites/CEREAL/Barley_cultivation-Norway_Presentation_2018.pdf" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_norway.jpg"></a> <a href="https://www.resilience.org/stories/2020-03-09/the-last-crop-before-the-desert/" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_morocco.jpg"></a> <a href="https://ipad.fas.usda.gov/highlights/2008/11/eth_25nov2008/" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_ethiopia.gif"></a> </div> <div class="column" style="max-width:44%"> <a href="https://kizilvest.ru/20150827-v-kizilskom-rajone-nachalas-uborochnaya-strada/" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_kizilskoye.jpg"></a> <a href="https://www.bloomberg.com/news/articles/2020-09-13/iraq-to-offer-first-ever-barley-exports-as-rains-yield-surplus" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_iraq.jpg"></a> <a href="https://thewire.in/agriculture/covid-19-lockdown-crop-harvest-farmers" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_rajasthan.jpg"></a> </div> <div class="column" style="max-width:8%; font-size: 15px;"> <p style="text-align: center; font-size: 25px; line-height: 1em;"> <strong> Cebada en todo el mundo </strong></p> <p>Tjøtta (Nordland, Noruega)</p> <p>Kiliskoye (Chelyabinsk, Rusia)</p> <p>Marchouch (Rabat, Marruecos)</p> <p>Bagdad (Bagdad, Irak) </p> <p>Aksum (Tigray, Etiopía) </p> <p>Hanumangarh (Rajasthan, India) </p> <p style="font-size:9px;line-height: 1em;">Creditos y detalles al dar click en cada imagen</p> </div> </div> --- # ¡Semillas con morfología diversa! <div class="row"> <div class="column" style="max-width:45%"> <a href="https://www.nationalgeographic.co.uk/travel/2020/05/photo-story-from-barley-fields-to-whisky-barrels-in-rural-scotland" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_seed_scotland.jpg"></a> </div> <div class="column" style="max-width:55%"> <a href="https://www.tibettravel.org/tibetan-culture/highland-barley.html" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_seed_tibet.jpg"></a> <a href="https://www.doi.org/10.1007/978-1-4419-0465-2_2168" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_historical_expansion.jpg"></a> <p style="font-size: 15px; line-height: 1em"> Cebada de Escocia y el Tibet. </p> <p style="font-size: 15px; line-height: 1em"> Expansión histórica de la cebada como cultivo. </p> <p style="font-size:9px;line-height: 1em;">Créditos y detalles al dar click en cada imagen</p> </div> </div> --- # Cebada: con nosotros desde el inicio <p style="font-size: 15px; text-align: left; color: DimGrey;"> Tabletas cuneiformes de Mesopotamia con espigas de cebada </p> <div class="row"> <div class="column" style="max-width:30%"> <img src="../figs/Ancient-Mesopotamia-tablet.jpg"> <img src="../figs/mesopotamian-tablet.jpg"> </div> <div class="column" style="max-width:35%"> <img src="../figs/beerancientegypt.jpg"> <p style="font-size: 15px; text-align: center; color: DimGrey;"> Registro egipcio del consumo de cerveza </p> </div> <div class="column" style="max-width:35%"> <img src="../figs/yoram_cave_barley.webp"> <p style="font-size: 15px; text-align: center; color: DimGrey;"> Cebada y arqueobotánica (Cueva Yoram, Israel. <a href="http://dx.doi.org/10.1038/ng.3611" target="_blank">Mascher et al 2016)</a></p> </div> </div> -- - La cebada es el 4to grano más cultivado, después del maíz, arroz y trigo. [(FAO 2020)](https://doi.org/10.4060/cb1993en) --- background-image: url("../figs/composite_hybrid_mixture.jpg") background-size: 400px background-position: 95% 5% # Cruce Compuesto II (CC II) <div class="row"> <div class="column" style="max-width:50%"> <img src="../figs/composite_cross_people.jpg"> <p style="font-size: 15px; color: DimGrey;"> Esquina superior izquierda: </p> <p style="font-size: 15px; text-align: center; color: DimGrey;"> Mary Martini, Harry Harlan, Coit Suneson, G.A. Wiebe, Robert Allard, Dan Koenig </p> </div> <div class="column" style="max-width:50%"> <img src="../figs/barley_world.jpg"> <p style="font-size: 15px; text-align: center; color: DimGrey;"> 28 sepas fundadoras </p> </div> </div> - Experimento iniciado en 1929 en Aberdeen, Idaho - En búsqueda del mejor cruce posible para California - Enfoque y mantenimiento en UC Davis y UC Riverside --- background-image: url("../figs/composite_cross_v_05.svg") background-size: 450px background-position: 95% 50% # Diseño del experimento .pull-left[ - 28 variedades fundadoras - Consideramos todos los posibles cruces de los fundadores ( `\(F_1\)` ) - Autopolinizamos cada uno de los híbridos ( `\(F_2\)` ) - Plantamos la progenie y les permitimos autopolinizarse - Vemos pasar casi 60 generaciones - Contamos con semillas de cada generación y linaje - Cuantificar la morfología - Secuenciar el ADN - ¿Cuáles genes fueron favorecidos? - ¿Cómo cambió la morfología? ] --- background-image: url("../figs/composite_cross_v_05.svg") background-size: 450px background-position: 95% 50% # Diseño del experimento .pull-left[ - **28 variedades** fundadoras - Consideramos todos los posibles cruces de los fundadores ( `\(F_1\)` ) - Autopolinizamos cada uno de los híbridos ( `\(F_2\)` ) - Plantamos la progenie y les permitimos autopolinizarse - Vemos pasar casi 60 generaciones - Contamos con semillas de cada generación y linaje - **Cuantificar la morfología** - Secuenciar el ADN - ¿Cuáles genes fueron favorecidos? - ¿Cómo cambió la morfología? ] --- class: center, middle, inverse # ~~2. Genética: Zigosidad~~ --- class: inverse, center, middle # 3. Procesamiento de imágenes ## Escanes 3D de rayos X --- background-image: url("../figs/barley_lab_composition.jpg") background-size: 750px background-position: 99% 99% # Tomografías 3D .pull-left[ Imágenes voxelizadas Resolución de ~30 micras En tandas de 4 ¡2Gb+ por cada escán! ] --- class: inverse, right background-image: url("../figs/barley_hpcc.png") background-size: 1300px background-position: 0% 0% # Procesamiento de imágenes _ad-hoc_ <div class="row"> <div class="column" style="max-width:12%; color: Yellow; font-size: 15px;"> <img src="../figs/S017_0_original.gif"> <p style="text-align: center; background-color: #808080;"> Original </p> </div> <div class="column" style="max-width:12%; color: Yellow; font-size: 15px;"> <img src="../figs/S017_1_normal.gif"> <p style="text-align: center; background-color: #808080;"> Estándar </p> </div> <div class="column" style="max-width:12%; color: Yellow; font-size: 15px;"> <img src="../figs/S017_2_unair.gif"> <p style="text-align: center; background-color: #808080;"> Limpio </p> </div> <div class="column" style="max-width:12%; color: Yellow; font-size: 15px;"> <img src="../figs/S017_3_denoise.gif"> <p style="text-align: center; background-color: #808080;"> Podado </p> </div> <div class="column" style="max-width:26%; color: Yellow; font-size: 15px;"> <img src="../figs/S015_alignment.jpg"> <p style="text-align: center; background-color: #808080;"> Etiquetado </p> </div> <div class="column" style="max-width:20%; color: Yellow; font-size: 15px;"> <img src="../figs/S019_L0_1.gif"> <p style="text-align: center; background-color: #808080;"> ¡Análisis! </p> </div> </div> -- - 224 escanes --- # Interés en la distribución de semillas <div class="row"> <div class="column" style="max-width:63%; padding: 0px 50px;"> <img src="../figs/seeds_batch1.png"> <img src="../figs/seeds_batch2.png"> </div> <div class="column" style="max-width:12%; color: Yellow; font-size: 15px; background-color: DarkRed; padding: 5px; border-radius: 15px 0px 0px 30px;"> <img src="../figs/seed_outlier1.png"> <img src="../figs/seed_outlier2.png"> <p style="text-align: right;"><strong> Semillas</strong></p> </div> <div class="column" style="max-width:12%; color: Yellow; text-align: center; font-size: 15px; background-color: DarkRed; padding: 5px;"> <img src="../figs/seed_outlier4.png"> <img src="../figs/seed_outlier6.png"> <p><strong>atípicas</strong></p> </div> <div class="column" style="max-width:12%; color: Yellow; text-align: center; font-size: 15px; background-color: DarkRed; padding: 5px; border-radius: 0px 15px 30px 0px;"> <img src="../figs/S102_1_44_0.png"> <img src="../figs/S129_0_37_0.png"> <p><strong> (Outliers) </strong></p> </div> </div> - `\(\sim38,000\)` semillas limpias. - Se vale descartar el 10% de la muestra. - Verificamos que las semillas atípicas no están concentradas. --- background-image: url("../figs/seed_mesh.png") background-size: 250px background-position: 80% 60% # Medidas tradicionales Todas las semillas son elipsoides: alineamiento basado en componentes principales .pull-left[ - Longitud - Altura - Ancho - Área superficial - Volumen - Superficie convexa - Volumen convexo  ] --- # Evolución morfológica y alometría <div class="row"> <div class="column" style="max-width:16%; color: Yellow; font-size: 15px;"> <img src="../figs/boxplot_all_length_v_es.png"> <img src="../figs/boxplot_all_height_v_es.png"> </div> <div class="column" style="max-width:16%; color: Yellow; font-size: 15px;"> <img src="../figs/boxplot_all_width_v_es.png"> <img src="../figs/boxplot_all_area_v_es.png"> </div> <div class="column" style="max-width:16%; color: Yellow; font-size: 15px;"> <img src="../figs/boxplot_all_vol_v_es.png"> <img src="../figs/boxplot_all_convexvol_v_es.png"> </div> <div class="column" style="max-width:16%; color: Yellow; font-size: 15px;"> <img src="../figs/boxplot_all_convexvolratio_v_es.png"> <img src="../figs/boxplot_all_convexarea_v_es.png"> </div> <div class="column" style="max-width:41%; color: Yellow; font-size: 15px;"> <img src="../figs/allometry_Length_vs_Vol.jpg"> <img src="../figs/allometry_Width_vs_Vol.jpg"> <img src="../figs/allometry_ConvexArea_vs_Vol.jpg"> </div> </div> --- class: inverse, middle, center # 4. Topología ## La Transformada de Característica de Euler (ECT) --- background-image: url("../../tda/figs/euler_characteristic_2.png") background-size: 450px background-position: 97% 50% # La característica de Euler .pull-left[ Figuras en 3D compuestas por `\(V_0\)` vértices, `\(V_1\)` arístas, y `\(V_2\)` caras. `\(\chi = V_0 - V_1 + V_2\)` **Números de Betti:** número de "agujeros" homológicamente distintos. - `\(\beta_0\)`: Número de componentes conexas - `\(\beta_1\)`: Número de ciclos - `\(\beta_2\)`: Número de agujeros / vacíos `\(\chi = \beta_0 - \beta_1 + \beta_2\)` En general: `\(\chi = \sum_{i=0}^n (-1)^iV_i = \sum_{i=0}^n (-1)^i\beta_i\)` ] --- class: center background-image: url("../../tda/figs/euler_characteristic_variety.jpg") background-size: 900px background-position: 50% 80% # Característica de Euler para formas diferentes La característica de Euler es un invariante topológico: invariante ante transformaciones suaves .pull-left[ Si la característica de Euler es **diferente**, los dos espacios/figuras son topológicamente distintos ] .pull-right[ Si los espacios son topológicamente distintos, la característica de Euler **puede ser igual.** ] --- # Complejos simpliciales - Vértices `\(\mathbf v_0, \ldots,\mathbf v_d\in\mathbb R^d\)` en posición general. -- - El **d-simplejo** es la envolvente convexa de los vértices. `$$S_d:=\left\{\sum_{i=0}^d\lambda_i\mathbf v_i\,:\,\lambda_i\geq0,\;\sum_{i=0}^d\lambda=1\right\}$$` <img src="../../tda/figs/simplices.svg" width="300px" style="display: block; margin: auto;" /> -- - Un complejo simplicial es un conjunto de simplejos bien pegados -- .pull-left[ <img src="../../tda/figs/complex-good.svg" width="250px" style="display: block; margin: auto;" /> - Bien pegados ] .pull-right[ <img src="../../tda/figs/complex-bad.svg" width="250px" style="display: block; margin: auto;" /> - Mal pegados ] --- # Complejos cúbicos - Conjuntos bien pegados de vértices, artistas, cuadrados y cubos de longitud unitaria. -- - Cubos de dimensión `\(0,1,2,3\)` respectivamente. -- - Pixels/voxels como vértices y vecinos deben de compartir una cara. -- <img src="../../tda/figs/binary_to_cubical_complex_es.svg" width="600px" style="display: block; margin: auto;" /> --- background-image: url("../figs/ecc_X.gif") background-size: 300px background-position: 90% 60% # Curva de característica de Euler (ECC) .pull-left[ - Un complejo simplicial/cúbico finito `\(M\subset\mathbb{R}^d\)` - Una dirección `\(\nu\in S^{d-1}\)` - La función de altura `\(f_{\nu}: M\to\mathbb R\)` - Definida como `\(f_{\nu}: x\mapsto\langle x,\nu\rangle\)`. - La filtración asociada `\(f_{\nu}^{-1}(-\infty,r\rbrack\simeq\{\Delta \in M : \langle x, \nu\rangle\leq r\:\forall \,x\in\Delta \}\)` - Definimos la curva de característica de Euler `\(\chi(M,\nu):\mathbb{R}\to\mathbb{Z}\)` `\(\chi(M,\nu)(r) = \chi(f_\nu^{-1}((-\infty,r\rbrack)).\)` ] --- background-image: url("../figs/ect.gif") background-size: 700px background-position: 50% 90% ## Transformada de característica de Euler (ECT) - Definimos `\(ECT(M): S^{d-1}\to\mathbb{Z}^{\mathbb{R}}\)` con `\(\nu\mapsto\chi(M,\nu)\)` - Concatenamos una cantidad infinita de ECCs. --- # ¿Porqué la transformada ECT? -- - Es fácil de calcular: una simple sucesión de sumas -- [**Teorema _(Turner, Mukherjee, Boyer 2014)_**](https://doi.org/10.1093/imaiai/iau011): La ECT es inyectiva para complejos simpliciales finitos en 3D -- [**Teorema _(ibid)_**](https://arxiv.org/abs/1310.1030): La ECT es una estadística suficiente para complejos simpliciales finitos en 3D -- _Traducción:_ - Dada todas las (infinitas) ECCs correspondientes a todas las direcciónes - Complejos simpliciales *distintos* corresponden a ECTs *distintas* - La ECT en efecto resume toda la información posible respecto a la forma de nuestro complejo -- Avances en reconstrucción práctica: construir un complejo simplicial a partir de una colección finita de ECCs. ([Turner, Curry](https://arxiv.org/abs/1805.09782), [Fasy](https://arxiv.org/abs/1912.12759), [Ghrist](https://doi.org/10.1007/s41468-018-0017-1)) --- background-image: url("../../tda/figs/sphere_partition.png") background-size: 280px background-position: 50% 90% # Idea de inyectividad - La demostración es constructiva - Escrita para la Transformada de Homología Persistente, `\(PHT(M)\)` - La idea clave consiste en calcular cambios de `\(\chi\)` a lo largo de la filtración `\(f_{\nu}\)` - Reconstruir `\(M\)` (con la cantidad mínima de vértices) - Vértices como puntos críticos de los diagramas de persistencia - Calcular los links de los vértices `\(x\)` para calcular las aristas y caras incidentes -- - [Prueba dibujística aquí.](../../tda/slides/lab200330.pdf) --- # Idea de inyectividad <embed src="../../tda/slides/lab200330.pdf" width="775px" height="475px" style="display: block; margin: auto;" type="application/pdf" /> --- class: inverse, center, middle # 5. Aprendizaje de máquina ## Resultados de ECT --- background-image: url("../../tda/figs/pole_directions_102.png") background-size: 350px background-position: 90% 50% # Calculando la ECT de cada semilla .pull-left[ - Con los ejes alineados, podemos asociar las mismas direcciónes a las mismas imágenes - 74 direcciones - 32 umbrales por dirección - Cada semilla es asociada a un vector `\(74\times32=2368\)` dimensional. 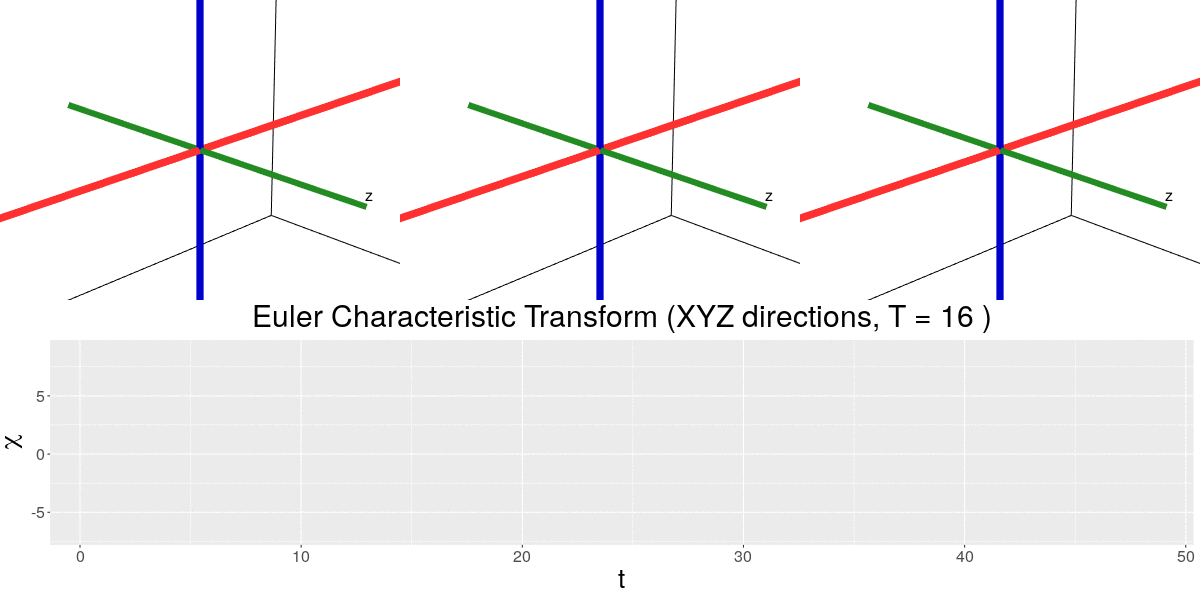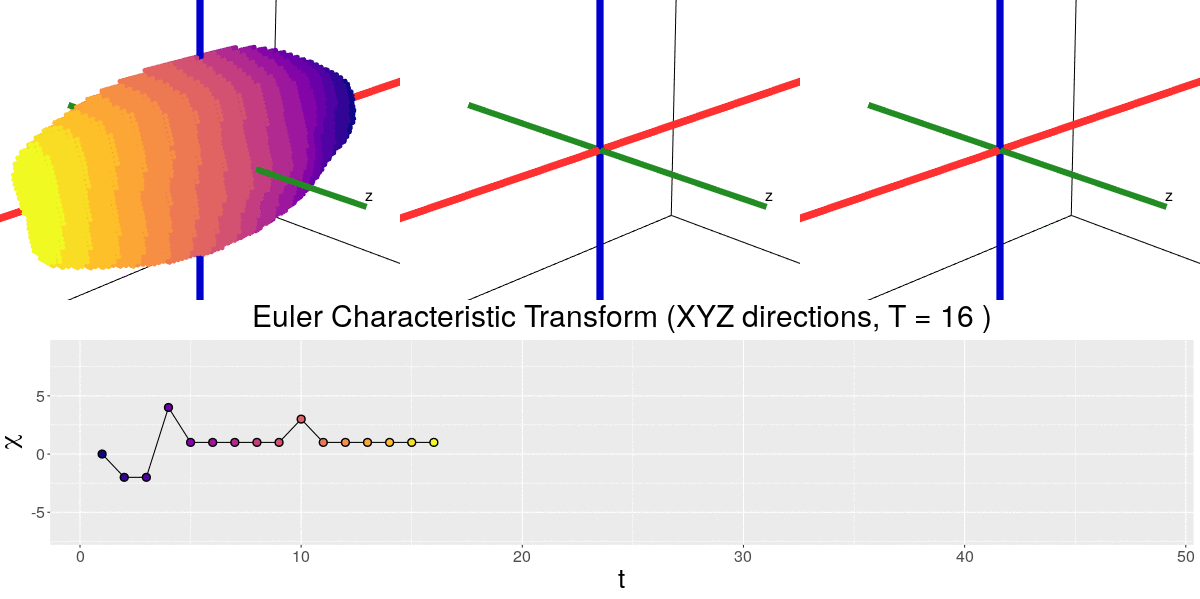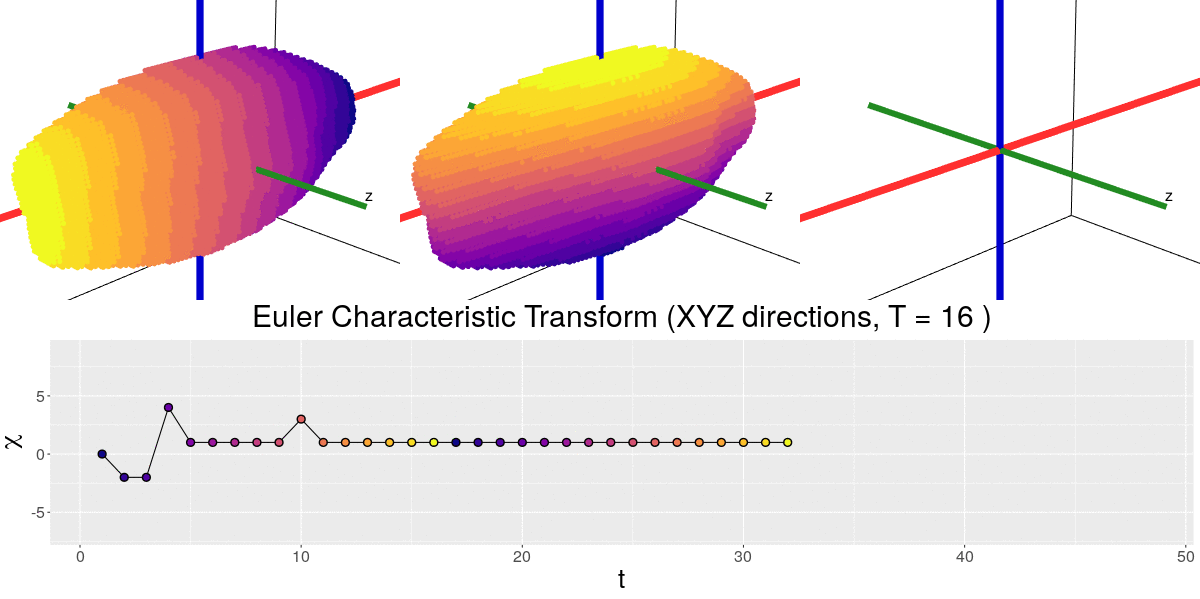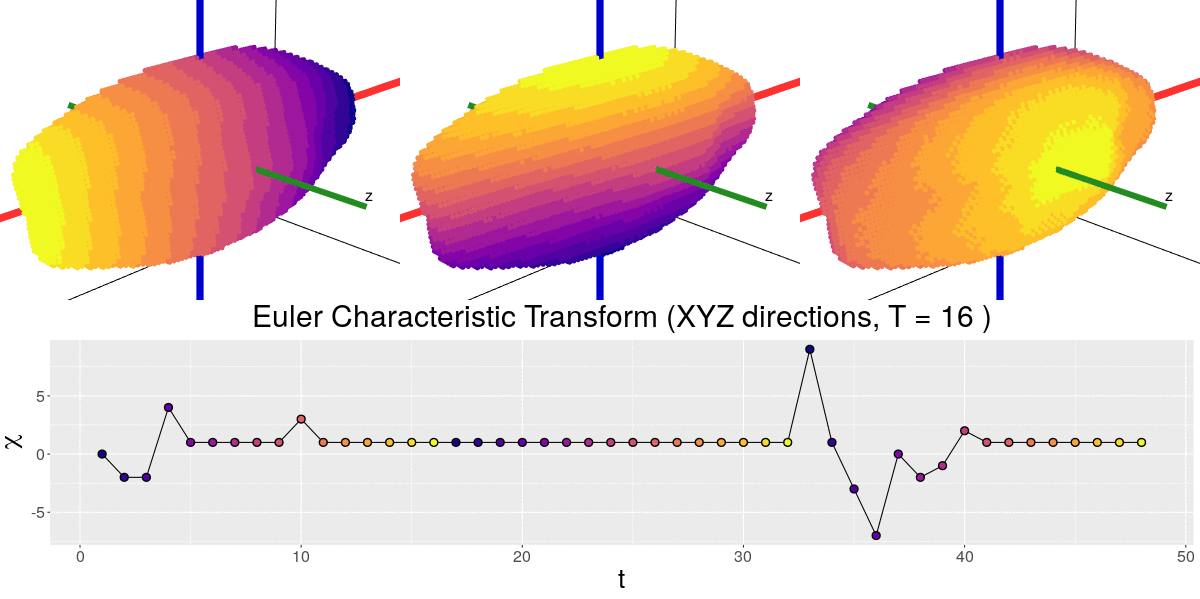 ] --- # Maldición de la Dimensión # 1 - Datos súper esparsos cuando la dimensión incrementa <img src="../../demat/figs/curse_of_dimensionality.svg" width="600px" style="display: block; margin: auto;" /> -- - Para cubrir el cubo unitario _d_-dimensional con `\(N\)` cubos sub-unitarios, éstos deben ser de longitud `\(d=\left(\frac1N\right)^{\frac1n}\)` -- - Como `\(d\to1\)` cuando `\(n\to\infty\)`, ¡podemos meter infinitos cubos unitarios dentro de un cubo unitario en dimensión infinita! -- - La intuición de _cercano_ y _lejano_ deja de funcionar. --- # Maldición de la Dimensión # 2 - Los puntos tienden a concentrarse en una esfera <img src="../../demat/figs/concentration-circle.svg" width="270px" style="display: block; margin: auto;" /> -- - Si contamos con `\(N\)` puntos `\(n\)`-dimensionales distribuidos de manera uniforme en el disco unitario, la media de la distancia `\(d\)` del origen al punto más cercano es `\(d = \left(1-\frac12^{\frac1N}\right)^{\frac1n}\)`. -- - Es más difícil hacer predicciones significativas cuando todos nuestros datos están en la frontera. --- background-image: url("../../demat/figs/pca_figure.jpg") background-size: 290px background-position: 99% 90% ## No supervisado: Principal Component Analysis (PCA) - Consideremos un datos `\(\mathbf x_1,\ldots,\mathbf x_n\in\mathbb R^d\)` (digamos `\(d=2368\)`) -- - Sea `\(1\leq k \leq d\)` fijo. Supongamos `\(k=2\)`. -- - Queremos hallar la _mejor_ aproximación afín `\(k\)`-dimensional `\(U\beta + \mu\)` tal que las columnas `\(U = [u_1, \ldots, u_k]\in\mathbb R^{d\times k}\)` forman una base ortonormal. -- - Problema de optimización: `$$\min_{\beta,\mu, U} \sum_{i=1}^n\|x_i - (U\beta_i + \mu)\|^2$$` tal que `\(U^\top U=I\)` y `\(\sum_i\beta_i=0\)`. -- - **Reducción de dimensiones** --- background-image: url("../../demat/figs/kernel_trick.png") background-size: 500px background-position: 50% 90% # Caso no lineal: Truco del kernel - Mapeamos nuestros datos a un espacio de dimensión alta donde su comportamiento sea lineal -- - `\(\Phi:\mathbb R^d\to\mathcal H\)`, con `\(\mathcal H\)` espacio de Hilbert de dimensión mayor. - Pensamos en una función **kernel** `\(K:\mathbb R^d\times\mathbb R^d\to\mathbb R\)`. `$$K(\mathbf{x,y})=\langle\Phi(\mathbf x), \Phi(\mathbf y)\rangle_{\mathcal{H}}$$` -- - El truco consiste en formular nuestro problema en términos de producto interno `\(K\)` y **nunca** lidiar directamente con `\(\mathcal H\)` ni `\(\Phi\)`. --- background-image: url("../../demat/figs/separable-svm.svg") background-size: 450px background-position: 50% 99% ## Supervisado: Support Vector Machines (SVM) - `\(n\)` puntos etiquetados `\(\{\mathbf x_i,y_i\}_{i=1}^n \subset \mathbb R^d\)` con `\(y_i\in\{-1,+1\}\)`. -- - Debemos hallar el __hiperplano H__ que los separa _mejor_, con `\(\mathbf H = \{\mathbf x\,:\,\langle\mathbf{x,w}\rangle+b=0\}\)`. -- - Problema de optimización `$$\begin{align} \min_{(\mathbf w,b)\in\mathbb R^d\times\mathbb R}\;\; &\frac12||\mathbf w||^2, \\ \textrm{tal que }\;\;& y_i(\langle \mathbf x_i,\mathbf w\rangle + b)\geq 1 \textrm{ para todo } i=1,\ldots,n. \end{align}$$` --- background-image: url("../figs/S012_L2_Blue_33.png") background-size: 150px background-position: 99% 50% # Metodología - **Meta:** Clasificar 28 variedades distintas de cebada basados únicamente en la morfología de sus granos - **3000** granos en total -- - Comparamos **3** conjuntos de descriptores morfológicos Descriptor | No. de descriptores -----------|-------------------- Tradicional | 11 Topológico (ECT + KPCA) | 12 Combinado (Trad + Topo) | 23 -- - Muestreamos sin remplazo 80% de semillas de cada sepa y entrenamos una SVM -- - Verificamos nuestra SVM con el 20% restante -- - Repetimos 50 veces y tomamos el promedio --- # Tradicionales vs Topológico .pull-left[ <img src="../figs/accuracy_traditional_rbfdot_12_founders.png" width="500px" style="display: block; margin: auto;" /><img src="../figs/confusion_matrix_traditional_crop.png" width="500px" style="display: block; margin: auto;" /> ] .pull-right[ <img src="../figs/accuracy_topological_rbfdot_12_founders.png" width="600px" style="display: block; margin: auto;" /><img src="../figs/confusion_matrix_topological_crop.png" width="600px" style="display: block; margin: auto;" /> ] --- # El tamaño sí importa <div class="row"> <div class="column" style="max-width:50%"> <img style="padding: 0 0 0 0;" src="../figs/boxplot_founders_Length.png"> <img style="padding: 0 0 0 0;" src="../figs/boxplot_founders_Area.png"> </div> <div class="column" style="max-width:50%"> <img style="padding: 0 0 0 0;" src="../figs/boxplot_founders_Width.png"> <img style="padding: 0 0 0 0;" src="../figs/boxplot_founders_Vol.png"> </div> </div> --- background-image: url("../figs/confusion_matrix_combined_crop.png") background-size: 500px background-position: 99% 70% # Pero si combinamos... .pull-left[ <img src="../figs/accuracy_combined_rbfdot_12_founders.png" width="500px" style="display: block; margin: auto;" /><img src="../figs/comparisonSVM_combined_vs_traditional_rbfdot_12_founders.png" width="500px" style="display: block; margin: auto;" /> ] --- # ...algo mejora 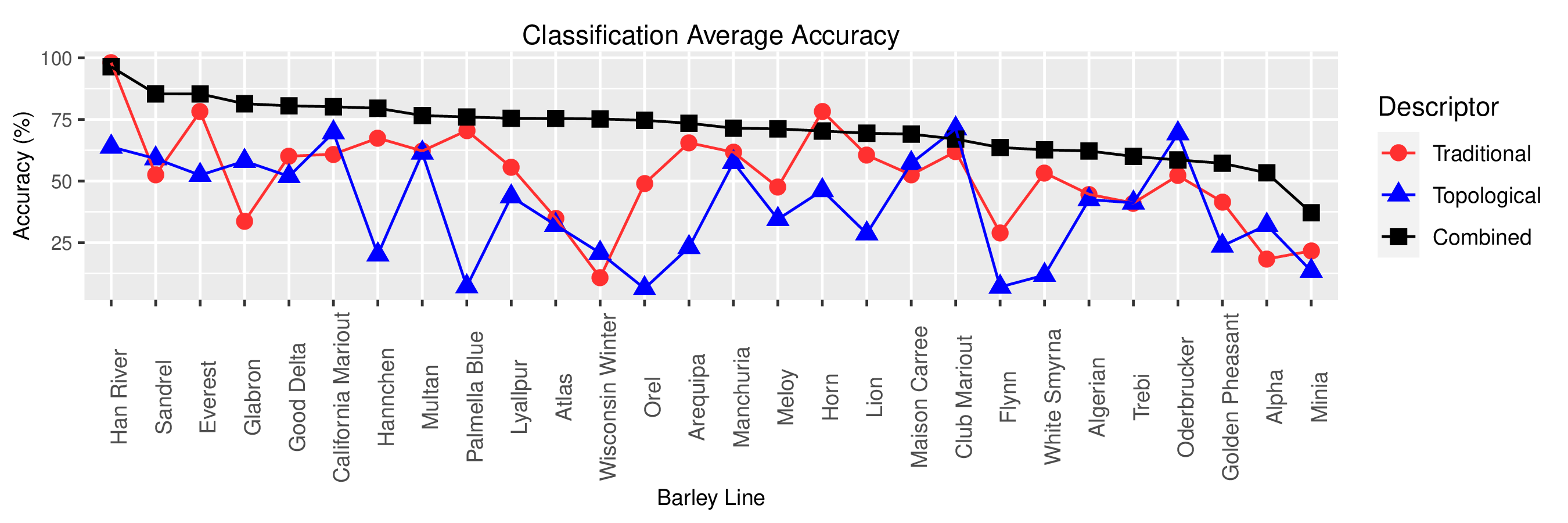 -- Descriptor | No. de descriptores | Precisión -----------|--------------------| ------- Tradicional | `\(11\)` | `\(51.9\% - 54.2\%\)` Topológico (ECT + KPCA) | `\(12\)` | `\(43.2\% - 45.7\%\)` Combinado (Trad + Topo) | `\(23\)` | `\(69.2\% - 71.9\%\)` --- class: inverse, center, middle # 6. Horizontes Genéticos ## Trabajo a futuro --- background-image: url("../figs/barley_domestication.jpg") background-size: 910px background-position: 50% 70% # Diversificación de la morfología floral --- background-image: url("../figs/BarleyEars_2_vs_6.jpg") background-size: 420px background-position: 0% 99% # Cebada de 2 vs 6 hileras <div class="row"> <div class="column" style="max-width:50%; margin-left:auto; margin-right:0;"> <p> El evento evolutivo de 2 vs 6 hileras es de suma importancia </p> <img src="../figs/barley_inflorescence.png"> <p style="font-size: 15px; color: DimGrey;"> <a href="http://dx.doi.org/10.1094/TQ-50-1-0331-01">Hertrich J. 2013</a>, <a href="https://doi.org/10.1016/j.jas.2013.09.015">Ros et al 2014</a> </p> </div> </div> <div class="row"> <div class="column" style="max-width:10%; margin-left:auto; margin-right:-350px;"> <img src="../figs/tworowbarley.png"> </div> <div class="column" style="max-width:10%; margin-left:auto; margin-right:50px;"> <img src="../figs/sixrowbarley.png"> </div> </div> --- # Genome Wide Association Study (GWAS) <img src="../figs/awn_gwas_a.jpg" width="1349" style="display: block; margin: auto;" /> - Ejemplo de [Milner et al 2019](https://doi.org/10.1038/s41588-018-0266-x) --- # Genome Wide Association Study (GWAS) <img src="../figs/awn_gwas_b.jpg" width="1368" style="display: block; margin: auto;" /> - Hallar los SNPs (Single Nucleotide Polymorphism) relevantes al rasgo --- # Genome Wide Association Study (GWAS) <img src="../figs/awn_gwas_c.jpg" width="1392" style="display: block; margin: auto;" /> -- - En principio, buscamos hallar los SNPs correspondientes a la forma --- class: inverse # Trabajo futuro - ¿Podemos usar la espiga entera? -- - Bailar la macarena con genética poblacional -- - Hallar un vínculo más concreto entre la variación morfológica y la variación genética -- - Si algo nos sobra, son imágenes botánicas <div class="row"> <div class="column" style="max-width:50%"> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/ikhuvGpJbeA?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> </div> <div class="column" style="max-width:50%"> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/a7JCIJRpF8U?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> </div> </div> --- class: right, bottom background-image: url("../figs/acknowledgments.jpg") background-size: 1000px background-position: 50% 40% Diapositivas disponibles en `https://bit.ly/cebada_fest` --- class: center, middle, inverse # 2. Genética: Zigosidad --- background-image: url("../../biology/figs/focus_DNA.jpg") background-size: 610px background-position: 97% 55% # Al final del día, todos somos ADN .left-column[ El ADN se compone de 4 nucleótidos: - Adenina(A) - Citosina(C) - Tiamina(T) - Guanina(G) Complementariedad - `\(A \leftrightarrow T\)` - `\(C \leftrightarrow G\)` Mecanismo natural de replicación ] --- background-image: url("../../biology/figs/mutant_phenotypes.jpg") background-size: 660px background-position: 97% 55% # Patrones hereditarios para un único gen .left-column[ **caraterística** = **propiedad** = **rasgo** **Fenotipo:** Forma tomada por un rasgo **Cruce:** Apareamiento controlado **Tipo silvestre:** El que se halla en la naturaleza **Mutante:** Organismo individual con alguna propiedad alterada ] --- background-image: url("../../biology/figs/alleles.jpg") background-size: 800px background-position: 50% 60% # Base molecular de patrones mendelianos **Alelo:** Variación de un gen conocido. Diferencia de uno o más nucléotidos **Mutación:** Cambio de nucleótidos en un alelo. --- background-image: url("../../biology/figs/mitosis_meiosis_d.jpg") background-size: 850px background-position: 50% 75% # Base cromática hereditaria de un único gen **Zigoto:** Huevo/óvulo fertilizado **Homozigoto:** Organismo con un par de alelos idénticos (línea pura: A/A ó a/a) **Heterozigoto:** Con diferente par de alelos (A/a) **Genotipo:** Combinación alélica (A/A, A/a, a/a) En principio, cada cromosoma tiene **la misma probabilidad** de ser heredado. --- background-image: url("../../biology/figs/mendel_single_gene_ratios.jpg") background-size: 790px background-position: 50% 75% # Combinatoria para un único alelo --- background-image: url("../../biology/figs/punnett_2_alleles_a.jpg") background-size: 370px background-position: 95% 35% # Misma idea para 2 alelos .pull-left[ - Razón fenotípica de 9:3:3:1 suponiendo independencia - La razón genotípica es mucho más distinta - *Recordatorio:* El ser dominante/recesivo no influencia la probabilidad de ser heredado - *Segunda Ley de Mendel:* Pares de genes distintos en **cromosomas distintos** se agrupan de manera independiente durante la formación de gametos.  ] --- background-image: url("../../biology/figs/tomato_varieties.jpg") background-size: 350px background-position: 90% 85% # Sintentizando líneas puras .pull-left[ - Si seguimos autopolinizando los heterozigotos, enventualmente tendremos progenie homozigótica. - Los homozigotos autopolinizados serán genotípicamente (y fenotípicamente) invariantes - Cada alelo tiene probabilidad `\(\frac12\)` de formar parte de una línea pura - No todos los homozigotos son viables ] .pull-right[ 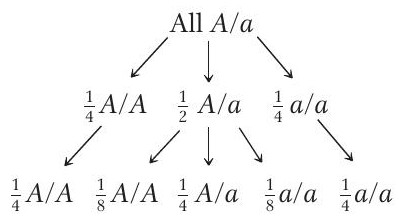 ]