class: center, middle, inverse, title-slide .title[ # Midiendo la forma en botánica ] .subtitle[ ## usando Análsis Topológico de Datos ] .author[ ### <strong>Erik Amézquita</strong>, Michelle Quigley, Tim Ophelders <br> Elizabeth Munch, Dan Chitwood <br> Dan Koenig, Jacob Landis <br> - ] .institute[ ### Computational Mathematics, Science and Engineering <br> Michigan State University <br> - ] .date[ ### 2021-12-15 <br> - <br> Slides: <a href="https://www.egr.msu.edu/~amezqui3/barley/slides/sembiomat_2021.html"><code>https://bit.ly/cebada_atd</code></a> ] --- background-image: url("../../img/endlessforms.png") background-size: 150px background-position: 89% 7% class: inverse # Morfología del reino vegetal <div class="row"> <div class="column" style="max-width:50%"> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/oM9kAq0PBvw?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/V39K58evWlU?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> </div> <div class="column" style="max-width:50%"> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/4GBgPIEDoa0?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/qkOjHHuoUhA?controls=0" frameborder="0" allow="accelerometer; autoplay; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> </div> </div> <p style="font-size: 24px; text-align: right; font-family: 'Yanone Kaffeesatz'">Mira más tomografías 3D en <a href="https://www.youtube.com/@endlessforms6756">youtube.com/@endlessforms6756</a></p> --- # Morfometría Tradicional <img src="https://nph.onlinelibrary.wiley.com/cms/asset/e0e8b362-efee-410a-a293-947c2d57acb9/nph16286-fig-0003-m.jpg" width="650" style="display: block; margin: auto;" /> <p style="font-size: 10px; text-align: right; color: Grey;"> Credits: <a href=" https://doi.org/10.1111/nph.16286">Gupta <em>et al.</em> (2019)</a></p> --- # Métodos morfométricos usuales no son suficientes .pull-left[ Descriptores Elípticos de Fourier (EFD)  <p style="font-size: 10px; text-align: right; color: Grey;"> Credits: <a href="https://doi.org/10.1016/j.cub.2016.02.033">Chitwood and Sinha (2016)</a></p> ] .pull-right[ Morfometría Geométrica (GMM)  <p style="font-size: 10px; text-align: right; color: Grey;"> Credits: <a href=" https://doi.org/10.1002/ppp3.10157">Chitwood (2020)</a></p> ] --- class: inverse # Necesitamos algo más robusto para datos más diversos <div class="row"> <div class="column" style="max-width:50%"> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/j0R9mMs5E50?controls=0" title="YouTube video player" frameborder="0" allow="accelerometer; autoplay; clipboard-write; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/2FmwkEA3tsY?controls=0" title="YouTube video player" frameborder="0" allow="accelerometer; autoplay; clipboard-write; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> </div> <div class="column" style="max-width:50%"> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/vxcSZsCs5BU?controls=0" title="YouTube video player" frameborder="0" allow="accelerometer; autoplay; clipboard-write; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> <iframe width="375" height="210" src="https://www.youtube-nocookie.com/embed/KXt-C-_OA3o?controls=0" title="YouTube video player" frameborder="0" allow="accelerometer; autoplay; clipboard-write; encrypted-media; gyroscope; picture-in-picture" allowfullscreen></iframe> </div> </div> <p style="font-size: 24px; text-align: right; font-family: 'Yanone Kaffeesatz'">Mira más tomografías 3D en <a href="https://www.youtube.com/@endlessforms6756">youtube.com/@endlessforms6756</a></p> --- # Análisis Topológico de Datos (ATD, TDA) <div class="row"> <div class="column" style="max-width:25%; font-size: 15px;"> <img style="padding: 25px 0 35px 0;" src="../figs/S019_L0_1.gif"> <p style="font-size: 25px; text-align: center; color: DarkRed;"> Datos </p> <ul> <li> Escanes rayos X </li> <li> Nubes de puntos </li> <li> Series de tiempo </li> <ul> </div> <div class="column" style="max-width:40%; padding: 0 25px 0 25px; font-size: 15px;"> <img src="../figs/ecc_X.gif"> <p style="font-size: 25px; text-align: center; color: DarkRed;"> Resumen Topológico </p> <ul> <li> Característica de Euler </li> <li> Diagramas de persistencia </li> <li> Mapper </li> <ul> </div> <div class="column" style="max-width:35%; font-size: 15px;"> <img src="../figs/svm_mds_ect.gif"> <p style="font-size: 25px; text-align: center; color: DarkRed;"> Análisis </p> <ul> <li> Estadística </li> <li> Aprendizaje de máquina </li> <li> Clasificación y predicción </li> <ul> </div> </div> --- class: inverse, middle, center # 1. Análisis Topológico de Datos ## La Transformada de Característica de Euler (ECT) --- # 1er ingrediente: Complejos - Nuestros datos como una colección de bloques elementales ( _células_ ) Vértices | Aristas | Caras | Volúmenes ---------|-------|-------|------- 0-dim | 1-dim | 2-dim | 3-dim - Una colección bien pegada de células: _complejo simplicial_ o _complejo cúbico_ - Contar el número de rasgos topológicos ( _agujeros_ ) Componentes conexas | Ciclos | Vacíos --------------------|--------|------- 0-dim | 1-dim | 2-dim .pull-left[ <img src="../../tda/figs/complex-good.svg" width="250px" style="display: block; margin: auto;" /> - 3 componentes, 1 ciclo, 1 vacío ] .pull-right[ <img src="../../tda/figs/cubical_complex_example.svg" width="250px" style="display: block; margin: auto;" /> - 2 componentes, 3 ciclos, 1 vacío ] --- # 2do ingrediente: Filtración - Asignamos un valor real cada simplejo y construimos el complejo poco a poco. - Observamos cómo cambia el número de rasgos topológicos a medida que crece el complejo .pull-left[ <img src="../figs/eigcurv_filter.gif" width="220px" style="display: block; margin: auto;" /><img src="../figs/gaussian_density_filter.gif" width="220px" style="display: block; margin: auto;" /> ] .pull-right[ <img src="../figs/eccentricity_filter.gif" width="220px" style="display: block; margin: auto;" /><img src="../figs/vrips_ver2.gif" width="220px" style="display: block; margin: auto;" /> ] -- - **Homología persistente** --- # Ejemplo 1 ## Detectar agujeros → detectar tejido cancerígeno .pull-left[  ] .pull-right[  ] <p style="font-size: 10px; text-align: right; color: Grey;"> Credits: <a href="https://doi.org/10.1016/j.media.2019.03.014">Qaiser <em>et al.</em> (2019)</a></p> --- # Ejemplo 2 ## Detectar agujeros → detectar recombinaciones .pull-left[  ] .pull-right[  ] <p style="font-size: 10px; text-align: right; color: Grey;"> Credits: <a href="https://doi.org/10.1073/pnas.1313480110">Chan <em>et al.</em> (2013)</a></p> --- # Ejemplo 3 ## Detectar agujeros → detectar conformaciones abiertas y cerradas .pull-left[   ] .pull-right[  ] <p style="font-size: 10px; text-align: right; color: Grey;"> Credits: <a href="https://doi.org/10.1515/sagmb-2015-0057">Kovacev-Nikolic <em>et al.</em> (2016)</a></p> --- class: inverse # Interludio comercial - Más información y referencias sobre la teoría matemática de ATD y sus aplicaciones a biología > Amézquita _et al._ (2020) "The shape of things to come: Topological data analysis and biology, from molecules to organisms". _Developmental Dynamics_ 249(7) pp. 816-833. DOI: [10.1002/dvdy.175](https://doi.org/10.1002/dvdy.175)  -- - Más herramientas de ATD: mapper, panoramas de persistencia, viñedos de persistencia, imágenes de persistencia, etc. --- # La característica de Euler `$$\chi = \#(\text{Vértices}) - \#(\text{Aristas}) + \#(\text{Caras}).$$` <img src="../../tda/figs/euler_characteristic_2.png" width="400" style="display: block; margin: auto;" /> - Resumimos todos los rasgos topológicos con la fórmula de Euler-Poincaré, `$$\chi = \#(\text{Componentes Conexas}) - \#(\text{Ciclos}) + \#(\text{Vacíos}).$$` - La característica de Euler es un **invariante topológico**. --- background-image: url("../figs/ecc_ver2.gif") background-size: 750px background-position: 50% 90% # Curva de Característica de Euler (ECC) - Consideramos un complejo `\(X\subset\mathbb{R}^d\)` - Una dirección unitaria `\(\nu\in S^{d-1}\)` - Y el subcomplejo que contiene todas las células debajo de la altura `\(h\)` respecto a la dirección `\(\nu\)` `$$X(\nu)_h =\{\Delta \in X\::\:\langle x,\nu\rangle\leq h\text{ for all }x\in\Delta\}$$` - La Curva de Característica de Euler de dirección `\(\nu\)` se define como la sucesión `$$\{\chi(X(\nu)_h)\}_{h\in\mathbb{R}}$$` --- background-image: url("../figs/ect_ver2.gif") background-size: 800px background-position: 50% 88% ## Transformada de Característica de Euler (ECT) - Repetimos para todas las direcciones posibles. - Más formalmente, la ECT es una función $$ `\begin{split} ECT(X):\; & S^{d-1} \to \mathbb{Z}^{\mathbb{R}}\\ &\nu\mapsto\{\chi(X(\nu)_h)\}_{h\in\mathbb{R}}. \end{split}` $$ --- # ¿Porqué la transformada ECT? -- - Es fácil de calcular: una simple sucesión de sumas -- [**Teorema _(Turner, Mukherjee, Boyer 2014)_**](https://doi.org/10.1093/imaiai/iau011): La ECT es inyectiva para complejos simpliciales finitos en 3D [**Teorema _(ibid)_**](https://arxiv.org/abs/1310.1030): La ECT es una estadística suficiente para complejos simpliciales finitos en 3D -- _Traducción:_ - Dada todas las (infinitas) ECCs correspondientes a todas las direcciónes - Complejos simpliciales *distintos* corresponden a ECTs *distintas* - La ECT en efecto resume toda la información posible respecto a la forma de nuestro complejo -- Desarrollar un algoritmo eficiente de reconstrucción de objetos en 3D a partir de una colección finita de direcciones sigue siendo un problema abierto ([Betthauser 2018](https://people.clas.ufl.edu/peterbubenik/files/Betthauser_Thesis.pdf); [Curry et al. 2018](https://arxiv.org/abs/1805.09782); [Fasy et al. 2019](https://arxiv.org/abs/1912.12759)). --- class: inverse, middle, center # 2. Cebada ## Cuantificando la morfología de la cebada usando la Característica de Euler ### Amézquita et al. (2021) [Measuring hidden phenotype: Quantifying the shape of barley seeds using the Euler Characteristic Transform](https://doi.org/10.1093/insilicoplants/diab033) --- class: inverse <div class="row"> <div class="column" style="max-width:44%"> <a href="https://kizilvest.ru/20150827-v-kizilskom-rajone-nachalas-uborochnaya-strada/" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_kizilskoye.jpg"></a> <a href="https://ipad.fas.usda.gov/highlights/2008/11/eth_25nov2008/" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_ethiopia.gif"></a> <a href="https://www.doi.org/10.1007/978-1-4419-0465-2_2168" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_historical_expansion.jpg"></a> </div> <div class="column" style="max-width:44%"> <a href="https://www.resilience.org/stories/2020-03-09/the-last-crop-before-the-desert/" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_morocco.jpg"></a> <a href="https://www.tibettravel.org/tibetan-culture/highland-barley.html" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_seed_tibet.jpg"></a> <a href="https://www.nationalgeographic.co.uk/travel/2020/05/photo-story-from-barley-fields-to-whisky-barrels-in-rural-scotland" target="_blank"><img style="padding: 0 0 0 0;" src="../figs/barley_seed_scotland_cropped.jpg"></a> </div> <div class="column" style="max-width:8%; font-size: 15px;"> <p style="text-align: center; font-size: 30px; line-height: 1em;"><strong>Cebada del mundo</strong></p> <p>Kiliskoye (Chelyabinsk, Rusia)</p> <p>Marchouch (Rabat, Marruecos)</p> <p>Aksum (Tigray, Etiopía)</p> <p>Salar (Tsetang, Tíbet)</p> <p>Expansión de la cebada</p> <p>Turriff (Aberdeenshire, Scotland)</p> <p style="font-size:9px;line-height: 1em;">Click on any picture for more details and credits</p> </div> </div> --- # Experimento Cross Composite II .pull-right[  ] --- background-image: url("../figs/composite_cross_v_01.png") background-size: 425px background-position: 95% 90% # Experimento Cross Composite II .pull-left[  - **28 fundadores/variedades** `\((F_0)\)` ] .pull-right[  ] --- background-image: url("../figs/composite_cross_v_02.png") background-size: 425px background-position: 95% 90% # Experimento Cross Composite II .pull-left[  - **28 fundadores/variedades** `\((F_0)\)` - Cruzamos `\({28 \choose 2}\)` **híbridos** `\((F_1)\)` ] .pull-right[  ] --- background-image: url("../figs/composite_cross_v_03.png") background-size: 425px background-position: 95% 90% # Experimento Cross Composite II .pull-left[  - **28 fundadores/variedades** `\((F_0)\)` - Cruzamos `\({28 \choose 2}\)` **híbridos** `\((F_1)\)` - **Auto-polinizar** los 379 híbridos resultantes ] .pull-right[  ] --- background-image: url("../figs/composite_cross_v_04.png") background-size: 425px background-position: 95% 90% # Experimento Cross Composite II .pull-left[  - **28 fundadores/variedades** `\((F_0)\)` - Cruzamos `\({28 \choose 2}\)` **híbridos** `\((F_1)\)` - **Auto-polinizar** los 379 híbridos resultantes - Cada línea crece en un área distinta en un terreno abierto. ] .pull-right[  ] --- background-image: url("../figs/composite_cross_v_05.svg") background-size: 425px background-position: 95% 90% # Experimento Cross Composite II .pull-left[  - **28 fundadores/variedades** `\((F_0)\)` - Cruzamos `\({28 \choose 2}\)` **híbridos** `\((F_1)\)` - **Auto-polinizar** los 379 híbridos resultantes - Cada línea crece en un área distinta en un terreno abierto **por 58 generaciones**. ] .pull-right[  ] --- ## Datos: rayos X → Procesamiento de imágenes <div class="row"> <div class="column" style="max-width:37.5%; color: Navy; font-size: 15px;"> <img style="padding: 2px 0 2px 0;" src="../figs/barley_packing.jpg"> <p style="text-align: center;">Cebada enviada desde California</p> </div> </div> --- ## Datos: rayos X → Procesamiento de imágenes <div class="row"> <div class="column" style="max-width:37.5%; color: Navy; font-size: 15px;"> <img style="padding: 2px 0 2px 0;" src="../figs/barley_packing.jpg"> <p style="text-align: center;">Cebada enviada desde California</p> </div> <div class="column" style="max-width:50%; color: Navy; font-size: 15px;"> <img src="../figs/x3000_setup.jpg"> <p style="text-align: center;">Reconstrucción de tomografías 3D ®</p> </div> </div> --- ## Datos: rayos X → Procesamiento de imágenes <div class="row"> <div class="column" style="max-width:37.5%; color: Navy; font-size: 15px;"> <img style="padding: 2px 0 2px 0;" src="../figs/barley_packing.jpg"> <p style="text-align: center;">Cebada enviada desde California</p> </div> <div class="column" style="max-width:50%; color: Navy; font-size: 15px;"> <img src="../figs/x3000_setup.jpg"> <p style="text-align: center;">Reconstrucción de tomografías 3D ®</p> </div> </div> <div class="row"> <div class="column" style="max-width:38%; color: Navy; font-size: 15px;"> <img style="padding: 2px 0 2px 0;" src="../figs/barley_xray_setup.jpg"> <p style="text-align: center;">Creatividad para escanear</p> </div> </div> --- ## Datos: rayos X → Procesamiento de imágenes <div class="row"> <div class="column" style="max-width:37.5%; color: Navy; font-size: 15px;"> <img style="padding: 2px 0 2px 0;" src="../figs/barley_packing.jpg"> <p style="text-align: center;">Cebada enviada desde California</p> </div> <div class="column" style="max-width:50%; color: Navy; font-size: 15px;"> <img src="../figs/x3000_setup.jpg"> <p style="text-align: center;">Reconstrucción de tomografías 3D ®</p> </div> </div> <div class="row"> <div class="column" style="max-width:38%; color: Navy; font-size: 15px;"> <img style="padding: 2px 0 2px 0;" src="../figs/barley_xray_setup.jpg"> <p style="text-align: center;">Creatividad para escanear</p> </div> <div class="column" style="max-width:17.5%; color: Navy; font-size: 15px;"> <img src="../figs/S019_L0_1.gif"> <p style="text-align: center;"> 975 espigas </p> </div> <div class="column" style="max-width:17%; color: Navy; font-size: 15px;"> <img src="../figs/S017_L3_1.gif"> <p style="text-align: center;"> 3 generaciones </p> </div> </div> --- ## Datos: rayos X → Procesamiento de imágenes <div class="row"> <div class="column" style="max-width:37.5%; color: Navy; font-size: 15px;"> <img style="padding: 2px 0 2px 0;" src="../figs/barley_packing.jpg"> <p style="text-align: center;">Cebada enviada desde California</p> </div> <div class="column" style="max-width:50%; color: Navy; font-size: 15px;"> <img src="../figs/x3000_setup.jpg"> <p style="text-align: center;">Reconstrucción de tomografías 3D ®</p> </div> </div> <div class="row"> <div class="column" style="max-width:38%; color: Navy; font-size: 15px;"> <img style="padding: 2px 0 2px 0;" src="../figs/barley_xray_setup.jpg"> <p style="text-align: center;">Creatividad para escanear</p> </div> <div class="column" style="max-width:17.5%; color: Navy; font-size: 15px;"> <img src="../figs/S019_L0_1.gif"> <p style="text-align: center;"> 975 espigas </p> </div> <div class="column" style="max-width:17%; color: Navy; font-size: 15px;"> <img src="../figs/S017_L3_1.gif"> <p style="text-align: center;"> 3 generaciones </p> </div> <div class="column" style="max-width:20.5%; color: Navy; font-size: 15px;"> <img src="../figs/S017_L0_seed_10_0.gif"> <p style="text-align: center;"> 38,000 semillas </p> </div> </div> --- ## Procesamiento de imágenes → Descriptores tradicionales .pull-left[ - Longitud - Ancho - Altura - Área de superficie - Volumen   ] -- .pull-right[    ] --- background-image: url("../figs/S012_L2_Blue_33.png") background-size: 150px background-position: 99% 50% # Juego y estrategia - **Meta:** Clasificar 28 variedades de cebada usando únicamente información morfológica de los granos. - **3121** granos en total -- .pull-left[ <img src="../figs/pole_directions_p7_m12_crop.jpg" width="150" style="display: block; margin: auto;" /> ] .pull-right[ - 158 direcciones - 16 rebanadas por dirección - Cada semilla es asociada a un vector `\(158\times16=2528\)`-dim - Dimensiones reducidas por separado con KPCA y UMAP ] -- - Comparamos **3** conjuntos de descriptores morfológicos Descriptor | No. of descriptors -----------|-------------------- Tradicional | 11 Topológico (ECT → KPCA/UMAP) | ~~2528~~ → 12 Combinado (Trad ⊕ Topo) | 23 -- - Muestreamos aleatoriamente 75/25 para entrenamiento y prueba. - Repetimos el muestreo y SVM 100 veces y consideramos el promedio. --- # Clasificación de 28 líneas con SVM <style type="text/css"> .tg {border-collapse:collapse;border-color:#93a1a1;border-spacing:0;margin:0px auto;} .tg td{background-color:#fdf6e3;border-bottom-width:1px;border-color:#93a1a1;border-style:solid;border-top-width:1px; border-width:0px;color:#002b36;font-family:Arial, sans-serif;font-size:14px;overflow:hidden;padding:10px 5px; word-break:normal;} .tg th{background-color:#657b83;border-bottom-width:1px;border-color:#93a1a1;border-style:solid;border-top-width:1px; border-width:0px;color:#fdf6e3;font-family:Arial, sans-serif;font-size:14px;font-weight:normal;overflow:hidden; padding:10px 5px;word-break:normal;} .tg .tg-2bhk{background-color:#eee8d5;border-color:inherit;text-align:left;vertical-align:top} .tg .tg-0pky{border-color:inherit;text-align:left;vertical-align:top} .tg .tg-gyvr{background-color:#eee8d5;border-color:inherit;font-size:100%;text-align:left;vertical-align:top} </style> <table class="tg"> <thead> <tr> <th class="tg-0pky">Descriptores</th> <th class="tg-0pky">No. de descriptores</th> <th class="tg-0pky">Precisión</th> <th class="tg-0pky">Recall</th> <th class="tg-0pky">F1</th> </tr> </thead> <tbody> <tr> <td class="tg-2bhk">Tradicional</td> <td class="tg-2bhk">11</td> <td class="tg-2bhk">0.57 ± 0.058</td> <td class="tg-2bhk">0.56 ± 0.019</td> <td class="tg-2bhk">0.55 ± 0.019</td> </tr> <tr> <td class="tg-0pky">Topológico</td> <td class="tg-0pky">12</td> <td class="tg-0pky">0.75 ± 0.047</td> <td class="tg-0pky">0.75 ± 0.016</td> <td class="tg-0pky">0.74 ± 0.016</td> </tr> <tr> <td class="tg-2bhk">Combinado</td> <td class="tg-2bhk">23</td> <td class="tg-2bhk">0.87 ± 0.031</td> <td class="tg-2bhk">0.86 ± 0.010</td> <td class="tg-2bhk">0.86 ± 0.010</td> </tr> </tbody> </table> <img src="../figs/avg_f1_combined_158_16_12_umap_horz.png" width="700" style="display: block; margin: auto;" /> --- # Descriptores morfológicos tradicionales  --- # Descriptores morfólogicos topológicos + UMAP 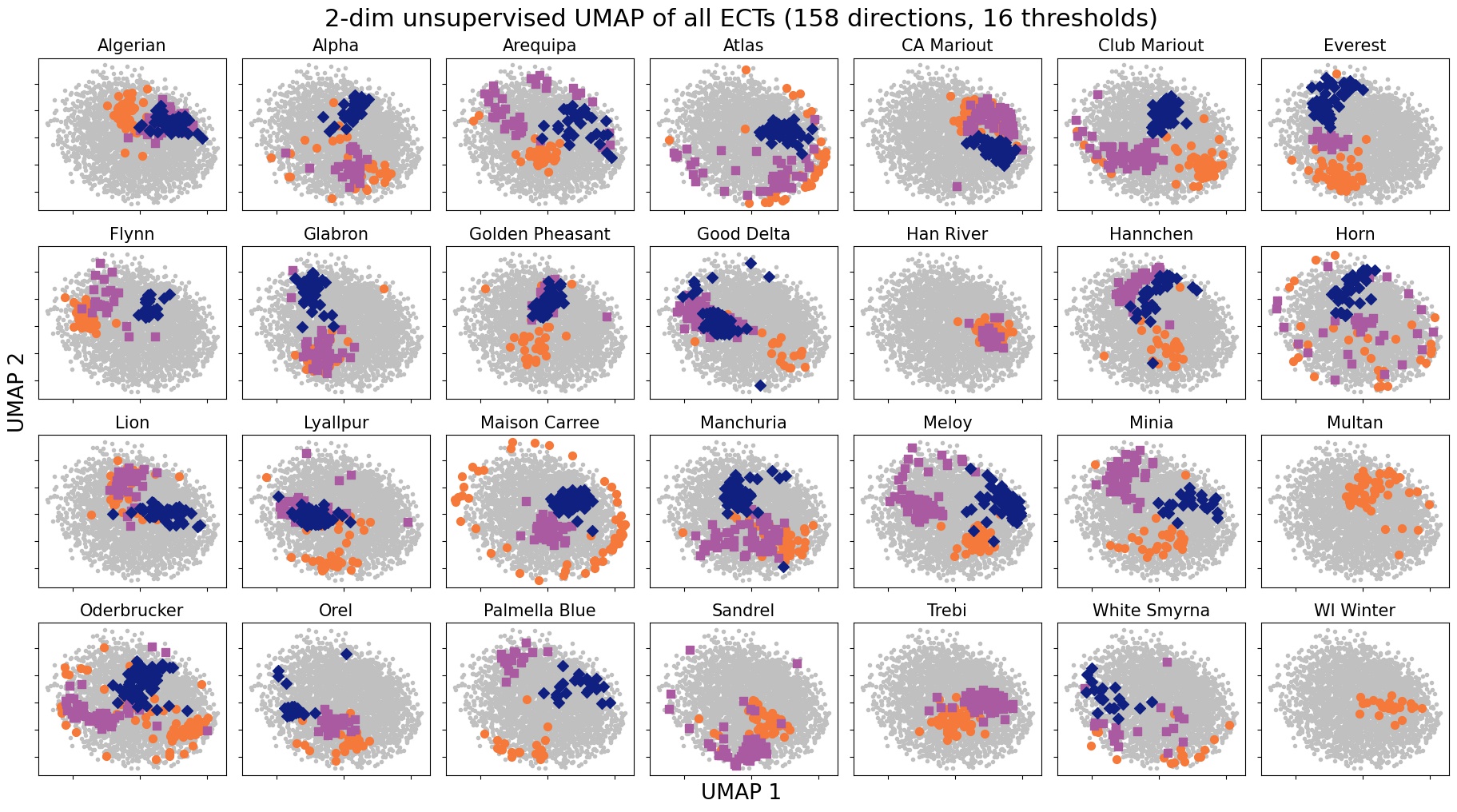 --- # Descriptores morfológicos topológicos + KPCA 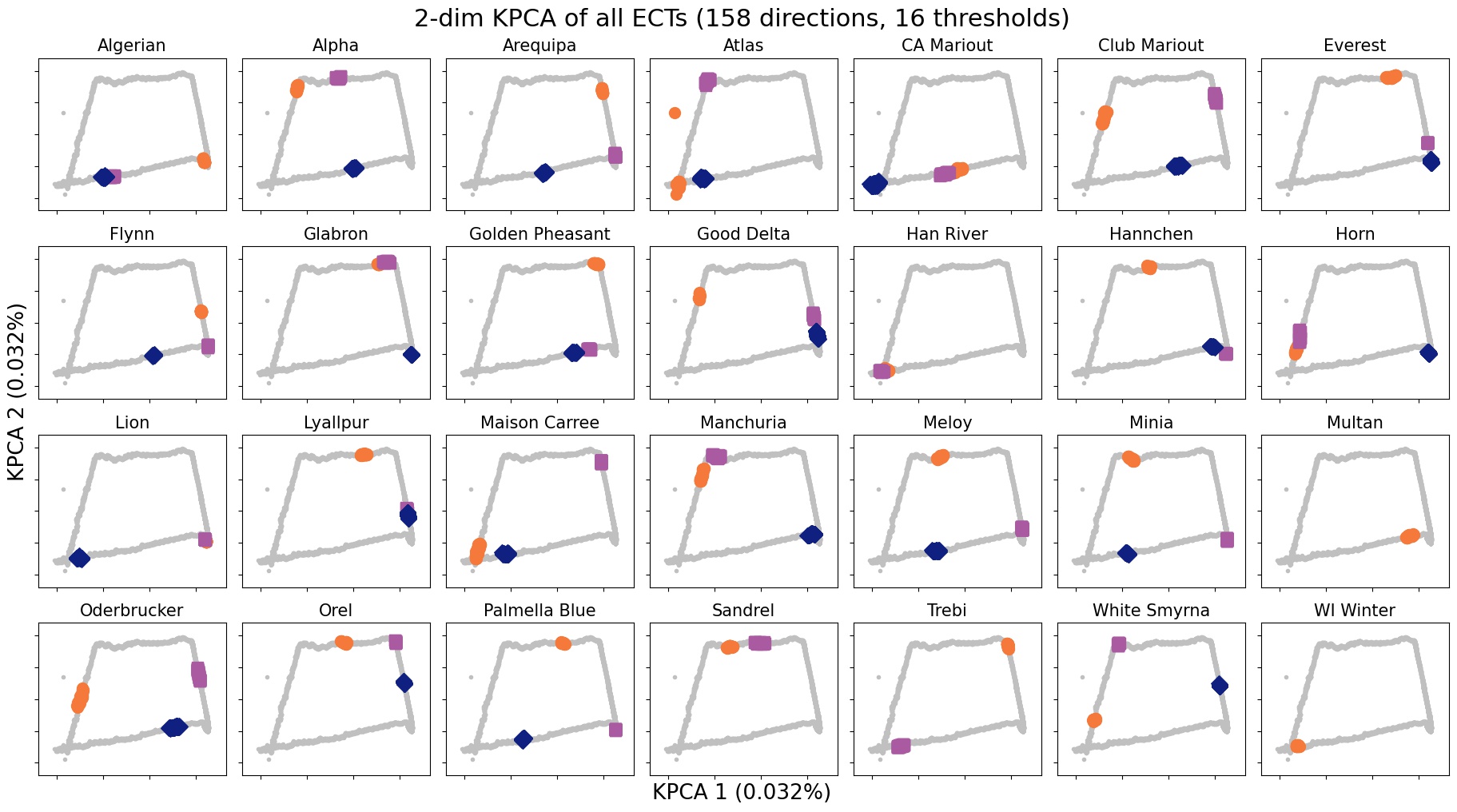 --- # Información topológica al descubierto .pull-left[ - Análisis de varianzar para determinar las direcciones y rebanadas más relevantes. - La hendidura central y la parte inferior de la semilla es bastante descriptiva! <img src="../figs/kruskal_wallis_topo_summary.jpg" width="300" style="display: block; margin: auto;" /> ] -- .pull-right[ <img src="../figs/discerning_directions.png" width="225" style="display: block; margin: auto;" /> 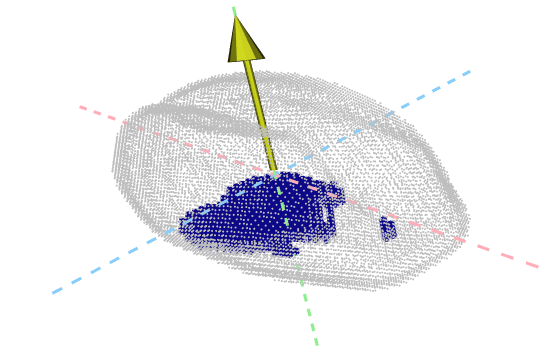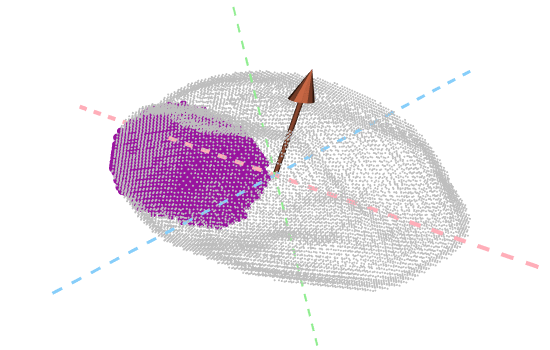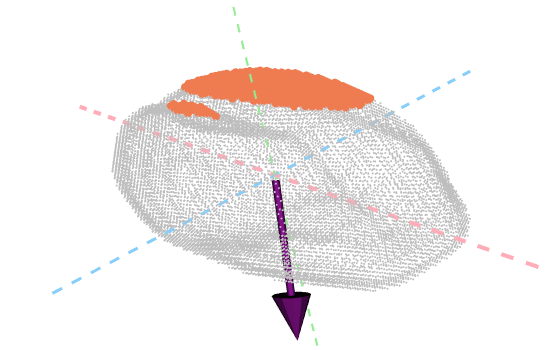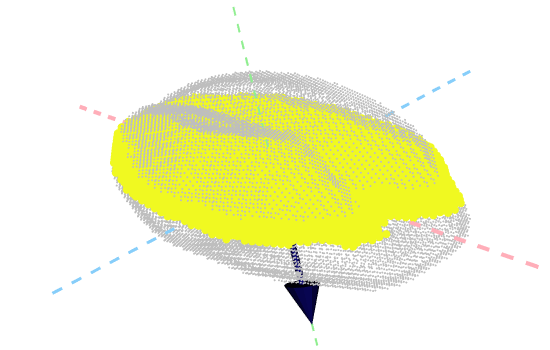 ] --- # Moviéndonos a terreno semi-supervisado - Entrenamos un SVM con el 100% de los fundadores `\((F_0)\)` - Clasificamos a la progenie `\((F_{18}\text{ and }F_{58})\)` para detectar enriquecimiento de genotipo. .pull-left[  ] .pull-right[  ] --- class: right, bottom, inverse background-image: url("../figs/acknowledgments.jpg") background-size: 1000px background-position: 50% 40% - [`https://bit.ly/cebada_atd`](https://www.egr.msu.edu/~amezqui3/barley/slides/sembiomat_2021.html)